Artículos de Revisión
Staphylococcus pseudintermedius y el enfoque de Una Salud
Staphylococcus pseudintermedius and the One Health approach
ANALECTA VETERINARIA
Universidad Nacional de La Plata, Argentina
ISSN: 1514-2590
Periodicidad: Semestral
vol. 40, núm. 2, 2020
Recepción: 08 Septiembre 2020
Revisado: 28 Octubre 2020
Aprobación: 04 Noviembre 2020

Resumen: El concepto de Una Salud se basa en la interdependencia entre la salud humana y la salud animal, vinculadas al medio ambiente en el que coexisten. Staphylococcus pseudintermedius es un microorganismo que forma parte de la microbiota de la piel y las mucosas en animales, especialmente en la especie canina de la que se suele aislar como agente etiológico de diferentes enfermedades. En los últimos años fue reconocido como un agente zoonótico. Si bien en condiciones normales no coloniza al hombre, puede causarle enfermedad bajo ciertas circunstancias. S. pseudintermedius cuenta con varios factores de virulencia, tales como enzimas, proteínas de superficie y toxinas, que participan en la adherencia, formación de biopelículas y evasión de la respuesta inmune. Posee resistencia múltiple a los agentes antimicrobianos, lo que dificulta el tratamiento de las infecciones que produce y, además, constituye un reservorio de genes de resistencia que pueden transmitirse a otras especies. Finalmente, los métodos convencionales usados en el laboratorio de diagnóstico no son suficientes para su identificación, por lo que muchas veces, no es posible confirmar su participación como agente etiológico de una enfermedad. El propósito de esta revisión fue exponer las características que permiten considerar a S. pseudintermedius en el marco de Una Salud.
Palabras clave: S pseudintermedius, virulencia, resistencia antimicrobiana, Una Salud.
Abstract: The interdependence between human and animal health linked to the environment in which they coexist are the bases of the One Health approach. Staphylococcus pseudintermedius is a skin and mucous membrane commensal in animals, especially in dogs, where it can cause different diseases. In recent years, S. pseudintermedius has been recognized as a zoonotic agent;although it does not colonize humans under normal conditions, it can cause disease under certain circumstances. Staphylococcus pseudintermedius has several virulence factors such as enzymes, surface proteins, and toxins that are involved in adherence, biofilm formation, and evasion of the immune response. In addition, it has multiple antimicrobial resistance which makes treatment difficult, and also constitutes a reservoir of resistance genes that can be transmitted to other species. Finally, conventional methods used in the diagnostic laboratory do not allow proper identification of S. pseudintermedius; therefore, in many occasions, diagnosis cannot be confirmed. The objective of this review was to describe S. pseudintermedius from a One Health approach.
Keywords: S pseudintermedius, virulence, antimicrobial resistance, One Health.
Introducción
El concepto “Una salud” fue introducido a comienzos del milenio, resumiendo en pocas palabras una noción conocida desde hace más de un siglo, es decir, que la salud humana y la sanidad animal son interdependientes y están vinculadas a los ecosistemas en los cuales coexisten. Con este párrafo inicial la Organización Mundial de Sanidad Animal (OIE) hace referencia en su página oficial sobre el significado de Una Salud (OIE, 2020) y desde 2010, junto con la Organización mundial de la salud (OMS) y la Organización de las naciones unidas para la alimentación y la agricultura (FAO), forma parte de la Alianza Tripartita para acordar un marco de referencia de colaboración (OIE, 2010). Según este enfoque, muchos son los temas que se pueden abordar por la interrelación entre ecosistema, salud animal y salud humana. Más aún, el intercambio hombre/animal evolucionó considerablemente y desde 2008 existen entidades públicas y privadas que exploran la interacción entre ellos (McCune et al., 2020). Esta concepción sobre el tema, está apoyada por otras instituciones tales como One Health Commission y One Health Initiative, las cuales adoptan el compromiso de educar a las futuras generaciones con esa perspectiva (Lueddeke et al., 2016). Algunas organizaciones internacionales, como el Clinical and Laboratory Standards Institute (CLSI), avalan esta iniciativa desde hace varios años, estableciendo estándares para bacterias del hombre, animales, agentes zoonóticos y antroponóticos utilizando las pruebas de sensibilidad antimicrobiana (Bobenchik et al., 2017).
Si bien hay numerosas zoonosis bacterianas que se abordan desde Una Salud, otras están emergiendo, como por ejemplo Staphylococcus pseudintermedius (S. pseudintermedius). Este coco grampositivo, microbiota de la piel y mucosas del perro y otras especies, es también un reconocido agente etiológico de enfermedades en la clínica veterinaria y está cada vez más asociado a infecciones en medicina humana (Bannoehr & Guardabassi, 2012) .
Varios son los factores involucrados en la transmisión de S. pseudintermedius desde los animales al hombre, así como también la relación con el medio en el que ambos habitan. El objetivo de esta revisión es dar a conocer cuáles son los componentes y las circunstancias que hacen que S. pseudintermedius sea una bacteria para ser considerada en el marco de Una Salud.
Taxonomía y hábitat de S. pseudintermedius
S. pseudintermedius pertenece a la familia Staphylococcaceae, género Staphylococcus(Schleifer & Bell, 2010).
El primer estudio en el que se describieron diferencias entre estafilococos coagulasa positivos de origen animal fue reportado por Hajek (1976) en aislamientos realizados a partir de palomas, perros, visones y caballos. En su trabajo a partir de pruebas bioquímicas y propiedades microbiológicas, describió una nueva especie de estafilococo a la que llamó Staphylococcus intermedius (S. intermedius). Posteriormente, y con la utilización de otras técnicas tales como la tipificación por fagos (Kawano et al., 1982), ribotipado (Hesselbarth & Schwarz, 1995) y análisis genético de la región intergénica de 16S 23S (Bes et al., 2002), se observó gran heterogeneidad de S. intermedius y se presumió la existencia de subespecies. Devriese et al. (2005), propusieron un nuevo taxón -S. pseudintermedius-, a partir de muestras tomadas de cuatro especies animales distintas, considerando sus características de crecimiento, resultados de pruebas bioquímicas e hibridación de ADN. La revisión de la clasificación por Sasaki et al. (2007) propone un grupo ´Staphylococcus intermedius group´ (SIG) en el cual incluye tres especies de estafilococos: S. intermedius, S. pseudintermedius y S. delphini.
En 2018 se incorpora al grupo SIG Staphylococcus cornubiensis, aislado a partir de una infección de piel humana (Murray et al., 2018) y más recientemente Staphylococcus ursi proveniente de osos negros (Ursus americanus) (Perreten et al., 2020).
Ross Fitzgerald (2009), en su revisión, remarca los aspectos de la reclasificación genética y afirma que S. pseudintermedius es la especie que predomina en caninos, mientras que S. intermedius lo es en otros animales.
S. pseudintermedius es la especie dentro del grupo SIG que habita como microbiota en la piel y mucosas del perro y se lo considera comensal y patógeno oportunista (Chrobak-Chmiel et al., 2018). Los datos recabados por Bannoehr & Guardabassi (2012) describen que constituye el 90 % de los cocos grampositivos en caninos y se distribuye principalmente en nariz, boca, periné e ingle.
En gatos clínicamente sanos, se reconoce su prevalencia, entre diferentes especies de estafilococos, como el coco que más predomina dentro de los coagulasa positivos de la piel (Lilenbaum et al., 1998).
En el humano está asociado con aquellas personas que tienen contacto estrecho con perros y gatos y comparten su hábitat. En estos casos, la bacteria puede adaptarse y, bajo la influencia de diversos factores, producir infecciones. (Kmieciak & Szewczyk, 2018). Puede encontrase también en otras especies animales (Devriese et al., 1985; Devriese et al., 2005).
Los estafilococos poseen la característica de permanecer en forma viable en el interior de los hogares, tanto en el polvo ambiental como en diferentes superficies, soportando ambientes secos entre tres semanas y tres meses (Beard-Pegler et al.,1988). La supervivencia depende de factores tales como la composición del polvo, la temperatura y humedad y la superficie de los materiales. La introducción de estas bacterias en los hogares se adjudica a los animales y personas (secreciones, estornudos, descamación epitelial ), fómites, alimentos y medio ambiente en general, mientras que el tiempo de permanencia de las mascotas y las personas dentro de los ambientes hogareños compromete la dinámica, exposición y transmisión (Davis et al., 2012).
Dificultades en la identificación en el laboratorio de diagnóstico microbiológico
La identificación de S. pseudintermedius en el laboratorio de diagnóstico microbiológico ofrece algunos inconvenientes. Eso se debe a que los métodos convencionales de cultivo e identificación mediante pruebas bioquímicas que se utilizan de rutina en la mayoría de los laboratorios de diagnóstico humano y veterinario no son suficientes para diferenciarlo de las especies que componen el grupo SIG y de otras especies de estafilococos coagulasa positivos.
Los sistemas comerciales con pruebas estandarizadas, tales como API Staph 32 (bio Mérieux), Rap-ID (Remel) y Staph-Zym (Rosco Diagnostica) tampoco resultan adecuados, según las investigaciones de Geraghty et al. (2013) sobre la eficacia de los citados métodos fenotípicos comerciales para el diagnóstico de estafilococos de origen animal. Las versiones automatizadas, tales como VITEK® Systems, BD PhoenixTM Automated Systems, TREK Sensititre ® Diagnostic Systems ™ dependen de la base de datos y, a menudo, no permiten discriminar entre S. pseudintermedius y Staphylococcus felis(Guardabassi et al., 2017).
Se han desarrollado diferentes estrategias moleculares para identificar S. pseudintermedius. Una de ellas es la detección del gen nuc mediante reacción en cadena de la polimerasa (PCR) múltiple desarrollada por Sasaki et al.(2010) para diferenciar cocos coagulasa positivos de origen animal. Este gen es altamente conservado en especies relacionadas, por lo que no resulta útil para identificar S. pseudintermedius si se usa PCR en tiempo real (RT-PCR). La técnica basada en la amplificación de genes mediante PCR seguida del estudio del polimorfismo de los fragmentos de restricción ha permitido diferenciar S. pseudintermedius de otros del grupo SIG. Los genes estudiados fueron el gen pta y el gen katA utilizando las enzimas de restricción MboI (Bannoehr et al., 2009) y TaqI (Blaiotta et al., 2010), respectivamente.
La secuenciación del ADN de 16S-rRNA utilizada frecuentemente para la identificación bacteriana, no es capaz de discriminar entre especies de estafilococos. El acceso de los laboratorios de investigación a la realización de la secuenciación completa de genomas (WGS), y la disponibilidad de las secuencias en las bases de datos, permitió encontrar regiones conservadas en el genoma de S. pseudintermedius como el gen spsJ, que no están presentes en otras especies de estafilococos. La amplificación de este gen mediante PCR pone a disposición una técnica de detección sensible y específica de especie (Verstappen et al., 2017).
El desarrollo de la tecnología MALDI-TOF MS permitió la utilización de la espectrometría de masas en la identificación de microorganismos mediante el análisis de proteínas, principalmente ribosomales, a través de la creación de un espectro de masas que es específico para cada género y especie. Decristophoris et al. (2011) describieron el poder de diferenciación del método con las especies que integran el grupo SIG.
En la revisión elaborada por el Grupo de Estudio de Microbiología Veterinaria (ESGVM), establecida dentro de la Sociedad Europea de Microbiología Clínica y Enfermedades Infecciosas (ESCMID), se describe, con algunas citas, las dificultades que presenta la identificación de especies de estafilococos en dermatología veterinaria. Se muestra el rendimiento de los métodos bioquímicos convencionales y automatizados para microorganismos involucrados en afecciones de la piel y se los compara con el MALDI-TOF. Así, concluyen que la identificación, mediante pruebas fenotípicas, de S. pseudintermedius, S. felis y Staphylococcus schleiferi es inadecuada y la única especie que permite ser identificada con certeza es Staphylococcus aureus. MALDI-TOF está calificado como bueno para S. aureus, S. felis y S. schleiferi (no distingue subespecies), mientras que para S. pseudintermedius la identificación es inadecuada usando la base de datos estándar, sin embargo es excelente cuando tiene incorporada la base de datos extendida (Guardabassi et al., 2017).
La dificultad en la identificación de S. pseudintermedius con técnicas disponibles en el laboratorio veterinario sigue siendo desfavorable e impacta en el tratamiento de las afecciones (inadecuada elección de agentes antimicrobianos en las pruebas para determinar su sensibilidad) y en la emergencia de los datos epidemiológicos que involucran a esta bacteria (Guardabassi et al., 2017).
Factores de virulencia
La capacidad de una bacteria de causar enfermedad en un huésped depende de su patogenicidad y de factores del huésped. La patogenicidad está dada por la capacidad de producir determinados factores de virulencia que favorecen la invasión, la degradación de tejidos y la evasión de la respuesta inmune.
S. pseudintermedius produce una variedad de factores de virulencia, que incluyen enzimas, proteínas de superficie y toxinas (Bannoehr & Guardabassi, 2012; Gómez-Sanz et al., 2011; Ross Fitzgerald, 2009). Algunos de estos factores intervienen en la adhesión de la bacteria a la superficie de las células epiteliales y a los componentes de la matriz extracelular (Tabla 1).
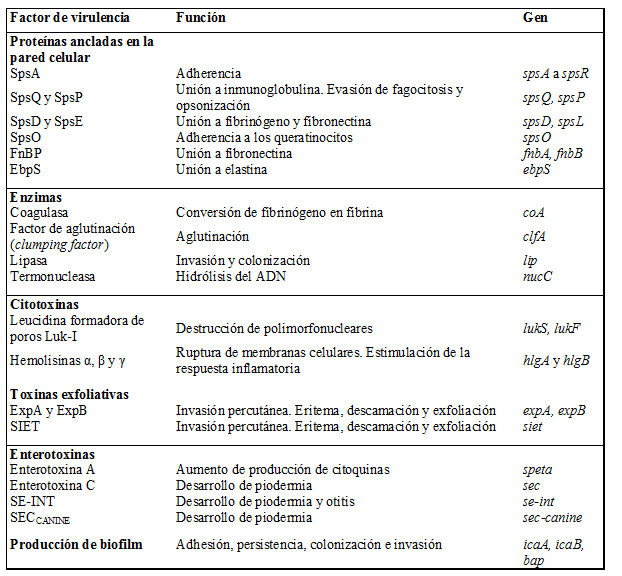
La coagulasa de S. pseudintermedius tiene propiedades multifuncionales que contribuyen a la evasión inmune. Se une a protrombina, inmunoglobulina y complemento, atrapando a las bacterias en una red de fibrina que las protege de la fagocitosis.
S. pseudintermedius tiene la capacidad de unirse a fibrinógeno, fibronectina y citoqueratina, lo que explica su capacidad de adherirse a los queratinocitos caninos (Geoghegan et al., 2009).
Una familia de proteínas de superficie ancladas en la pared celular conocidas como CWA (por sus siglas en inglés cell wall-anchored proteins), interviene en la adherencia de la bacteria a las proteínas de la matriz extracelular del huésped y participa del proceso de internalización de S. pseudintermedius. Estas proteínas están codificadas por, al menos, dieciocho genes presentes en el genoma de S. pseudintermedius (spsA a spsR).
S. pseudintermedius también expresa proteínas de superficie como factor de aglutinación (clumping factor) y una proteína de unión a inmunoglobulina conocida como proteína A (SpA), similares a las de S. aureus. Los aislamientos de S. pseudintermedius que expresan la proteína A pueden evadir la opsonización y la fagocitosis, lo que lleva a infecciones persistentes (Grandolfo, 2018). Está codificada por los genes spsQ y spsR, el primero de ellos presente en todos los aislamientos de S. pseudintermedius y el segundo asociado a ciertos linajes (Balachandran et al., 2018).
La unión al fibrinógeno y la fibronectina está mediada por SpsD y SpsL. La SpsD también está involucrada en la unión a la citoqueratina, que es uno de los principales constituyentes de la piel de los mamíferos, y a la elastina (Bannoehr et al., 2011). En suero de perros con piodermia se encontró IgG específica para SpsD y SpsL, demostrando que ambas proteínas se expresan durante la infección (Bannoehr et al., 2011) Otra proteína Sps importante involucrada en la colonización del huésped es la SpsO, que interviene en la adherencia a los queratinocitos caninos (Bannoehr & Guardabassi, 2012).
Un estudio comparativo de la fisiopatología de S. pseudintermedius y otras especies del género sobre infecciones osteoarticulares, mostró que S. pseudintermedius es capaz de adherirse a la fibronectina, de internalizarse y de persistir en los osteoblastos, incluso en mayor medida que S. aureus(Maali et al., 2016).
Las leucocidinas formadoras de poros dañan el sistema inmune innato, la primera línea de defensa contra estos patógenos. S. pseudintermedius produce una leucotoxina conocida como Luk-I que está presente en el 90 % de las cepas aisladas en clínica veterinaria y es muy similar a la PVL (Panton-Valentine leukocidin) de S. aureus (Futagawa-Saito et al., 2004a). Luk-I está codificada por dos genes cotranscriptos lukS y lukF y presenta actividad leucotóxica sobre polimorfonucleares.
S. pseudintermedius produce α-hemolisina y β-hemolisina y causa hemólisis de eritrocitos de conejo y de oveja a distintas temperaturas (Bannoehr & Guardabassi, 2012).
Las modulinas solubles en fenol (PSM) son una familia de péptidos anfipáticos con múltiples funciones en la patogénesis estafilocócica. Pueden causar la lisis de muchos tipos de células humanas, incluidos leucocitos y eritrocitos, estimular respuestas inflamatorias y contribuir al desarrollo de biopelículas (Cheung et al., 2014). Las PSM parecen relacionarse con el estilo de vida comensal de los estafilococos, facilitando su crecimiento y propagación en las superficies epiteliales. Las infecciones necrotizantes y purulentas se relacionan con la alta citotoxicidad observada luego de la invasión de S. pseudintermedius en las células del huésped.
Las toxinas exfoliativas estafilocócicas facilitan la invasión bacteriana percutánea produciendo eritema, descamación y exfoliación en caninos. Varias de estas toxinas están presentes en S. pseudintermedius.
La similitud entre las características clínicas de la piodermia canina y las del síndrome de piel escaldada por estafilococos en humanos, llevó a especular si S. intermedius también produce una toxina exfoliativa como factor de virulencia en la patogénesis de la piodermia canina. En un primer estudio realizado con 54 perros con piodermia no se logró detectar la toxina exfoliativa (Hesselbarth et al., 1994). Sin embargo, en un estudio realizado posteriormente en Japón se describió por primera vez la toxina SIET (Terauchi et al., 2003). La misma fue identificada en la mitad de los S. pseudintermedius aislados de infecciones de piel y otitis externa en Alemania (Lautz et al., 2006).
Desde entonces se comenzaron a descubrir diferentes toxinas exfoliativas llamadas Exi, que presentaban homología significativa con algunas toxinas exfoliativas de S. aureus (Futagawa-Saito et al., 2009). Se describió una codificada por el gen expB, presente en el 23 % de los aislamientos clínicos de piodermia superficial canina (Iyori et al., 2010) y, como consecuencia, los autores propusieron cambiar el nombre de Exi a ExpA, de acuerdo con la segunda toxina exfoliativa ExpB (Iyori et al., 2010). Estas toxinas ExpA y ExpB se encontraron también en hisopados nasales de perros sanos y de sus propietarios (Walther et al., 2012).
S. pseudintermedius también presenta enterotoxinas, como SECCANINE y SE-INT, que causan piodermia y otitis crónica (Edwards et al., 1997; Futagawa-Saito et al.,b). La SEC de S. pseudintermedius fue clonada, purificada, caracterizada y comparada con las de otras especies y, sobre la base de su asociación con infecciones caninas, se denominó SECCANINE (Edwards et al., 1997). La prevalencia de SECCANINE en la población de S. pseudintermedius es variable. Becker et al.(2001) detectaron la enterotoxina en el 12,6 % de los 247 aislamientos realizados. Por el contrario, en Japón, sobre un total de 44 aislamientos caninos se halló solo una cepa positiva para SECCANINE, aunque se identificó una nueva enterotoxina SE-INT, la que se detectó en todos los aislamientos (Futagawa-Saito et al.,2004b). En el mismo país, sobre 74 cepas de S. pseudintermedius aisladas de piodermia u otitis crónica, se halló SECCANINE en el 24 % de los aislamientos y SIET en el total de los mismos (Yoon et al., 2010). En Túnez, sobre 55 aislamientos de S. pseudintermedius sensibles a meticilina aislados de hisopados nasales de perros sanos, se detectó el gen seccanine en el 1,8 % (Gharsa et al., 2013), coincidentemente con lo observado en otros estudios realizados en perros sanos (Gómez-Sanz et al., 2011; Gómez-Sanz et al., 2013). En este estudio también se encontró que los genes lukS/F-I, siet y se-int estaban presentes en todos los aislamientos, mientras que el gen expA se halló solo en tres (Gharsa et al., 2013).
Otra característica importante en la patogénesis de las infecciones y la capacidad de sobrevivir y persistir en el ambiente que presenta esta especie es la de formar biopelículas. Se demostró que prácticamente todos los aislamientos de S. pseudintermedius las producen, por lo que se considera una característica esencial para su patogenicidad (Singh et al., 2013). El gen icaA que forma parte del operón ica desempeña un rol crucial en la producción de las mismas en S. pseudintermedius(Couto et al., 2015). Esta característica es importante en el desarrollo de infecciones crónicas y podría complicar el tratamiento de infecciones debido a la disminución de la eficacia de los agentes antimicrobianos (Osland et al., 2012).
La diversidad de genes de virulencia presente en los aislamientos de esta especie parece indicar que los mismos podrían desempeñar un papel importante en la gravedad de las infecciones y su potencial de causar enfermedad. Son, junto a otros factores, los que le permiten establecerse en el huésped de una especie en particular y de bacterias comensales convertirse en patógenos oportunistas.
Resistencia antimicrobiana
S. pseudintermedius frecuentemente presenta resistencia a diversos antibióticos. Es ampliamente resistente a las penicilinas lábiles a la penicilinasa, como la penicilina G, la ampicilina o la amoxicilina. Estudios realizados en Europa y Estados Unidos en S. pseudintermedius aislados de caninos y felinos revelaron que el gen blaZ responsable de la resistencia a la penicilina estaba presente en el 99 % de las cepas resistentes a meticilina (Kadlec et al., 2010; Perreten et al.,2010), como así también en aislamientos sensibles a meticilina (Kadlec & Schwarz, 2012).
La resistencia a meticilina en S. pseudintermedius (MRSP) está mediada por el gen mecA que codifica una proteína fijadora de penicilinas alterada (PBP2a) con baja afinidad por todos los antibióticos β-lactámicos. Esta resistencia es de amplio espectro, y confiere resistencia a todos los antibióticos β-lactámicos, excepto ceftarolina y ceftobiprole (Lakhundi & Zhang, 2018). El gen mecA se localiza en un elemento genético móvil conocido como SCCmec o casete cromosómico, que puede transferirse entre estafilococos de la misma o diferente especie (Black et al., 2009). Los elementos SCCmec encontrados en S. pseudintermedius, si bien muestran cierto grado de homología con los de S. aureus y otras especies de estafilococos, presentan gran diversidad (Perreten et al., 2013).
Los primeros aislamientos de S. intermedius resistentes a meticilina se detectaron fenotípicamente a principio de los años ‘80 en perros sanos y con piodermia (Pellerin et al., 1998), pero los primeros aislamientos confirmados mediante la detección del gen mecA fueron reportados en Estados Unidos en 1999 y en Europa en 2005 (Gortel et al., 1999; Loeffler et al., 2007; Moodley et al., 2014). Sin embargo, la emergencia de S. pseudintermedius resistente a la meticilina como un patógeno significativo en animales ocurrió en la década del 2000 (Weese & van Duijkeren, 2010). En los años siguientes su prevalencia aumentó notablemente en caninos, transformándose en un problema creciente para la medicina veterinaria (Bannoehr & Guardabassi, 2012; van Duijkeren et al., 2011; Weese & van Duijkeren, 2010).
La mayoría de los laboratorios de diagnóstico veterinario utilizan métodos fenotípicos para la detección de resistencia a la meticilina en estafilococos. El disco de cefoxitina usado para detectar resistencia a meticilina en otras especies de estafilococos no es adecuado para su detección en esta especie. Por ese motivo, en 2016 el CLSI incorporó puntos de corte de oxacilina para S. pseudintermedius y estableció que, en esta especie, ni el disco de cefoxitina ni la concentración inhibitoria mínima (CIM) de cefoxitina son apropiados para detectar resistencia a meticilina mediada por el gen mecA.
Una característica distintiva de S. pseudintermedius es que, tanto los aislamientos resistentes a meticilina como los sensibles, presentan una alta tasa de resistencia a antibióticos no β-lactámicos como aminoglucósidos, fluorquinolonas, lincosamidas, macrólidos, tetraciclinas, cloranfenicol y trimetoprima-sulfametoxazol (Kadlec & Schwarz, 2012; Moodley et al., 2014; Vigo et al., 2015).
Un estudio multicéntrico realizado en Canadá, Estados Unidos, Dinamarca, Alemania, Francia, Italia, Suecia, Suiza y Holanda sobre 103 aislamientos de MRSP no relacionados epidemiológicamente, encontró que, además de la resistencia a meticilina, presentaban resistencia a once antibióticos no β-lactámicos (Perreten et al., 2010).
La resistencia a macrólidos, lincosamidas y estreptograminas en estafilococos puede deberse a modificación del sitio de acción, eflujo activo del antibiótico o inactivación enzimática. El mecanismo más frecuente en S. pseudintermedius es MLSB codificado por el gen ermB, característico de otras especies de Gram positivos como enterococos y estreptococos (Guardabassi et al., 2004; Kadlec & Schwarz, 2012). En menor medida se encontraron otros genes de resistencia, como el gen lnuA, que codifica una lincosamida nucleotidil transferasa y el gen msrA que codifica una bomba de eflujo (Kadlec & Schwarz, 2012).
Los mecanismos de resistencia a tetraciclina en estafilococos son dos. El primero es la protección ribosomal del sitio de unión del antibiótico, codificado por los genes tetM y/o tetO, que confieren resistencia cruzada a tetraciclina, doxiciclina y minociclina. El segundo mecanismo es la presencia de bombas de eflujo, codificadas por los genes tetK y/o tetL, que expulsan activamente el antibiótico, mecanismo que confiere resistencia a tetraciclina y doxiciclina pero no a minociclina (Foster, 2017). En S. pseudintermedius, el gen más comúnmente detectado es el tetM (Kadlec & Schwarz, 2012). Sin embargo, los aislamientos de algunos clones particulares como el CC71 presentan con más frecuencia el gen tetK, por lo que minociclina conserva actividad en estos casos.
La resistencia a aminoglucósidos se debe principalmente a la acción de enzimas modificadoras codificadas por distintos genes. En S. pseudintermedius, los más prevalentes son aph(3@)-IIIa que confiere resistencia a kanamicina y neomicina, aac(6’)-Ie–aph(2’’)-Ia que confiere resistencia a gentamicina, tobramicina, kanamicina y amicacina y ant(4’)-Ia que confiere resistencia a tobramicina y kanamicina (Descloux et al., 2008).
La enzima cloranfenicol acetiltransferasa codificada por los genes cat constituye el principal mecanismo de resistencia a cloranfenicol en esta especie.
La resistencia a trimetoprima-sulfametoxazol en S. pseudintermedius se debe principalmente a mutaciones en el gen dfrG que codifica para una dehidrofolato reductasa (Gold et al., 2014; Norström et al., 2009).
Es muy frecuente que los aislamientos de S. pseudintermedius presenten resistencia a fluorquinolonas por alteración del sitio de acción de estas drogas. Esta alteración se debe a la acumulación de mutaciones puntuales en los genes grlA y grlB de la ADN girasa, y gyrA y gyrB de la topoisomerasa IV, que generalmente se producen en una región específica denominada región determinante de resistencia a quinolonas o QRDR. En S. pseuditermedius, las sustituciones aminoacídicas detectadas con mayor frecuencia son S80I y D84N en el gen grlA y S84L y E88G en el gen gyrA(Gagetti et al., 2019; Kadlec & Schwarz, 2012).
Si bien la resistencia a rifampicina no es frecuente en S. pseudintermedius, se encontraron aislamientos resistentes con mutaciones en la región determinante de resistencia a rifampicina del gen rpoB (Corro et al., 2018; Kadlec et al., 2011). Hasta el presente, no se detectó resistencia a glucopéptidos en S. peudintermedius.
La prevalencia de resistencia a cada familia de antibióticos varía de acuerdo con la región geográfica y con la población estudiada, pero la resistencia múltiple es una característica de la especie.
Los aislamientos que presentan resistencia a por lo menos tres familias de antibióticos se denominan multirresistentes (MDR) y los que conservan sensibilidad a dos clases de antibióticos o menos son extensamente resistentes (XDR) (Magiorakos et al., 2011). Algunas cepas de S. pseudintermedius son MDR y XDR, presentando resistencia a todos los antibióticos de administración oral utilizados para tratar infecciones en perros (Perreten et al., 2010). Se encontraron aislamientos resistentes a todos los antibióticos autorizados en medicina veterinaria, lo que lleva a considerar que sea una necesidad potencial emplear, en veterinaria, agentes antimicrobianos que solo están autorizados para medicina humana (Weese & van Duijkeren, 2010).
Las altas tasas de resistencia que presentan algunas de estas cepas hacen que en Suecia las infecciones producidas por MRSP se consideren una enfermedad animal de declaración obligatoria (SWEDRES-SVARM, 2015).
Potencial zoonótico de S. pseudintermedius
Según las OIE, el 60 % de las enfermedades humanas infecciosas son zoonosis y cada año aparecen cinco nuevas enfermedades humanas, tres de las cuales son de origen animal.
La transmisión de microorganismos entre especies animales y el hombre depende de numerosos factores, entre ellos las barreras que separan ambas especies. Gortazar et al., (2014) cita, por ejemplo, cuál es la barrera entre especies que determina la naturaleza y el nivel de exposición humana a patógenos zoonóticos y cuál es la barrera intrahumana que determina la capacidad de patógenos zoonóticos para infectar a un humano y hacer frente, de forma eficaz, a la respuesta inmunitaria del nuevo hospedador.
La primera barrera, que involucra la exposición humana al patógeno zoonótico, es relevante al considerar a S. pseudintermedius. Durante los últimos 50 años aumentó sustancialmente la cantidad de animales de compañía en la sociedad moderna y, al mismo tiempo, se produjo un cambio en el rol social de las mascotas, lo que ha generado un contacto más estrecho con los humanos (Pomba et al., 2017). Este contacto cercano entre los animales de compañía y los humanos brinda excelentes oportunidades para la transmisión entre especies de bacterias resistentes y sus genes de resistencia (Pomba et al., 2017).
La mera exposición a un agente no implica la transmisión y colonización de una especie a otra. Hay numerosos componentes involucrados además de los factores de virulencia ya descriptos, como el tropismo, la presencia de receptores celulares adecuados, la composición de la microbiota y el estado inmune del nuevo hospedador. Kmieciak & Szewczyk (2018), estudiaron algunos de los factores que contribuyen a la colonización de humanos con S. pseudintermedius y la posible adaptación al mismo. Encontraron que S. pseudintermedius, además de formar biopelículas y portar genes de virulencia que codifican para promover la infección, fue capaz de inhibir la microbiota residente de la piel humana como barrera que la protege de la infección. Varios autores reportaron el potencial zoonótico de esta especie, tanto en cepas sensibles como resistentes a meticilina (Börjesson et al., 2015; Ference et al., 2019; Somayaji et al., 2016; Talan et al., 1989; Vandenesch et al., 1995; van Duijkeren et al., 2008) y si bien el huésped preferido por S. pseudintermedius es el perro, son capaces de provocar infecciones invasivas y no invasivas en humanos.
En el primer caso de infección humana reportado en 2006 no se pudo establecer la relación del paciente con ninguna mascota, por lo que se interpretó como una infección comunitaria (van Hoovels et al., 2006). En el primer caso de infección por S. pseudintermedius resistente a la meticilina (MRSP), aislado de bacteriemia asociada a catéter en un paciente con adenocarcinoma gástrico, tampoco pudo conocerse si el paciente había tenido o no contacto con animales al momento de la infección (Campanile et al., 2007).
La primera vez que se comprobó trasmisión zoonótica fue en Estados Unidos en 2009, cuando un MRSP aislado de infección posquirúrgica de senos paranasales fue comparado con un aislamiento recuperado del perro del paciente, que había presentado episodios recientes de piodermia, y ambos aislamientos presentaron un patrón de PFGE similar (Kempker et al., 2009). También en 2009, en Suiza, se aisló MRSP de una infección posquirúrgica recurrente en un paciente operado de senos paranasales que convivía con un perro enfermo (Stegmann et al., 2010). En 2013, en Italia, se recuperó MRSP de una infección de herida de un paciente previamente sometido a trasplante de médula ósea que convivía con un perro (Savini et al., 2013). Otro estudio realizado en Canadá entre 2013 y 2015 reportó 24 casos de infección por S. pseudintermedius en humanos, tres de ellos MRSP. Dos de los aislamientos estudiados provenían de infección invasiva, una bacteriemia y una infección de prótesis articular, y, los demás, de infección de piel y partes blandas. Más del 90 % de los pacientes informaron haber estado en contacto con perros al momento de la infección (Somayaji et al., 2016). En España, entre 2014 y 2015, se reportaron cuatro casos de infección en humanos por S. pseudintermedius, tres de los cuales pudieron relacionarse, mediante PFGE y MLST, con las cepas recuperadas de los perros de los pacientes (Lozano et al., 2017). En un estudio retrospectivo realizado en California, Estados Unidos, entre 2007 y 2017, se encontraron 33 casos de infección de senos paranasales producidos por S. pseudintermedius (27 % de ellos MRSP), el 97 % de los pacientes convivían con perros (Ference et al., 2019).
En nuestro país, la primera vez que se recuperó un MRSP multirresistente de un paciente humano fue en 2017, aislado de una muestra de hueso, y la paciente refirió no haber estado en contacto con mascotas (Gagetti et al., 2020).
Afecciones en animales
Como otras especies de estafilococos, S. pseudintermedius es un patógeno oportunista en animales. Se lo describe como agente etiológico de enfermedades especialmente de piel en caballos (Devriese et al., 1985) gatos (Lilenbaum, et al., 1998) y perros (Bannoehr & Guardabassi, 2012). Sin embargo, es la especie canina en la que predomina como agente causal de las piodermias superficiales y profundas (Loeffler & Lloyd, 2018). En ella se lo considera microbiota y no causa enfermedad a menos que la barrera cutánea sea alterada por factores predisponentes, como dermatitis atópica, procedimientos médicos y quirúrgicos o trastornos inmunosupresores. La condición del huésped es crucial en la patogenia de la infección por esta bacteria (Bannoehr & Guardabassi, 2012). Algunos autores, diferencian la colonización de S. pseudintermedius en transitoria, intermitente y persistente, considerando a los animales que tienen colonización persistente como más propensos a la enfermedad por presentar mayor carga bacteriana (Hartmann et al., 2005).
Los caninos con dermatitis atópica son portadores de S. pseudintermedius en una mayor proporción que los individuos sanos (Fazakerley et al., 2009). También se observó una mayor adherencia a los queratinocitos en regiones de piel inflamada que en áreas no inflamadas (McEwan et al., 2006).
Los queratinocitos tienen función de barrera cutánea y en su fase final de diferenciación producen queratina, lípidos, ácidos grasos y ceramidas que los hacen permanecer activos (Momota et al., 2016). La capacidad de adhesión a los queratinocitos podría relacionarse con la diferente afinidad de este agente en las distintas especies animales (Simou et al., 2005). Así, la menor frecuencia con la que se registran afecciones en la piel en los gatos podría deberse a una menor adherencia de S. pseudintermedius a los queratinocitos felinos que a los caninos. Esto explicaría porqué los gatos son menos propensos a las piodermias por este agente, considerando a la adherencia como primer paso para la colonización e infección en la piel (Woolley et al., 2008).
El S. pseudintermedius también está relacionado con las otitis caninas, y la capacidad de formación de biopelículas es una característica que se destaca por incrementar su persistencia y llevar a fracasos en el tratamiento (Moreira et al., 2012). En equinos, se registró como agente etiológico de piodermia profunda en un animal de 8 años (Santos et al., 2008). Como patógeno oportunista, es una bacteria que se aísla a partir de hisopados óticos y vaginales, orina, líquido pericárdico, secreción conjuntival y lavado nasal de caninos (Vigo et al., 2015) y en muestras clínicas de gatos, conejos, leche de vaca y gerbiles (Chrobak et al., 2011; Wettstein et al., 2008).
Discusión
“Un Mundo, Una Salud”, describe el surgimiento de un nuevo concepto que subraya la súbita toma de conciencia colectiva del vínculo existente entre las enfermedades animales y la salud pública. Así lo describió la dirección de la OIE en 2009 (Vallat, 2009).
La construcción de este concepto evolucionó en el tiempo, de acuerdo con la visión del conocimiento y percepción de los términos salud /enfermedad hasta la perspectiva actual (Gavidia Catalán & Talavera, 2012).
Los componentes y las circunstancias que hacen a S. pseudintermedius una bacteria para abordarla con ese sentido se describieron en cada uno de los apartados anteriores; sin embargo, se destacan algunos aspectos.
La resistencia múltiple de S. pseudintermedius representa un desafío para el tratamiento de las infecciones que produce. Además, la dificultad para su identificación fenotípica en laboratorios clínicos y su similitud con S. aureus hacen que los métodos convencionales utilizados en la actualidad no sean lo suficientemente precisos. Es por eso que muchas veces no es posible confirmar su participación como agente etiológico de una enfermedad. Además, por este motivo, se producen errores en la interpretación de las pruebas de sensibilidad. La incorporación de la espectrometría de masa y las técnicas moleculares hizo que numerosos aislamientos identificados como S. aureus o S. intermedius fueran reclasificados como S. pseudintermedius.
La resistencia múltiple y la virulencia pueden influenciarse mutuamente. En muchos casos el costo energético que representa la adquisición y el mantenimiento de los mecanismos de resistencia se asocia con una disminución de la virulencia. Sin embargo, también hay evidencias que muestran lo contrario, ya que los elementos genéticos móviles que diseminan genes de resistencia muchas veces contienen genes de virulencia en el mismo elemento. Esto permite la selección de bacterias virulentas en condiciones de presión antibiótica y la selección de bacterias resistentes a los antibióticos en condiciones de infección (Linares-Rodríguez & Martínez-Menéndez, 2005).
La coexistencia de genes de virulencia y de resistencia que presenta esta especie comensal hacen que sean potenciales patógenos altamente virulentos y resistentes.
Se suma a este contexto el rol social de las mascotas y el contacto cada vez más estrecho con los humanos (Pomba et al., 2017).
Por otra parte, los veterinarios tienen mayores posibilidades de exponerse a S. pseudintermedius en su práctica y ambiente profesional (hospitales y clínicas). Weese (2012) reseña los principios generales de prácticas y métodos de control de S. aureus resistentes a la meticilina que se aplican en medicina humana y, luego, los relaciona con las medidas prácticas de control de infecciones que deberían implementarse en medicina veterinaria para limitar la propagación de estafilococos en pacientes, propietarios y personal veterinario.
La Asociación Nacional de Veterinarios de Salud Pública Estatal (NASPHV) describe los lineamientos generales para la prevención de enfermedades zoonóticas en el personal veterinario (Williams et al.,2015). En la misma línea la Federación de Asociaciones Veterinarias de Animales de Compañía Europeas (FECAVA) recomienda la higiene y el control de infecciones en la práctica veterinaria para prevenir y evitar la transmisión de infecciones nocosomiales a animales y humanos dentro del ámbito veterinario y en la comunidad (FECAVA, 2018).
En conclusión, S. pseudintermedius debe ser considerado en el marco de Una Salud y representa un problema para la salud pública, tanto por las infecciones que causa en animales y humanos, como por el reservorio de genes de resistencia y su potencial transferencia entre aislamientos de origen humano, animal y medio ambiente.
Declaración de conflicto de intereses.
No existe conflicto de intereses, incluyendo entre estos últimos las relaciones financieras, personales o de otro tipo con otras personas u organizaciones que pudieran influir de manera inapropiada en el trabajo.
Referencias
Balachandran M, Bemis DA, Kania SA. 2018. Expression and function of protein A in Staphylococcus pseudintermedius. Virulence. 9(1):390-401. https://doi.org/10.1080/21505594.2017.1403710
Bannoehr J, Ben Zakour NL, Reglinski M, Inglis NF, Prabhakaran S, Fossum E, Smith DG, Wilson GJ, Cartwright RA, Haas J, Hook M, van den Broek AH, Thoday KL, Fitzgerald JR. 2011. Genomic and surface proteomic analysis of the canine pathogen Staphylococcus pseudintermedius reveals proteins that mediate adherence to the extracellular matrix. Infection and Immunity. 79(8):3074-86. https://doi.org/10.1128/IAI.00137-11
Bannoehr J, Franco A, Iurescia M, Battisti A, Fitzgerald JR. 2009. Molecular diagnostic identification of Staphylococcus pseudintermedius. Journal of Clinical Microbiology. 47(2):469-71. https://doi.org/10.1128/JCM.01915-08
Bannoehr J, Guardabassi L. 2012. Staphylococcus pseudintermedius in the dog: taxonomy, diagnostics, ecology, epidemiology and pathogenicity. Veterinary Dermatology. 23:253-e52. https://doi.org/10.1111/j.1365-3164.2012.01046.x
Beard-Pegler MA, Stubbs E, Vickery AM. 1988. Observations on the resistance to drying of staphylococcal strains. Journal of Medical Microbiology. 26(4):251-5. https://doi.org/10.1099/00222615-26-4-251
Becker K, Keller B, von Eiff C, Brück M, Lubritz G, Etienne J, Peters G. 2001. Enterotoxigenic Potential of Staphylococcus intermedius. Applied and Environmental Microbiology. 67(12):5551-7. https://doi.org/10.1128/AEM.67.12.5551-5557.2001.
Bes M, Saidi Slim L, Becharnia F, Meugnier H, Vandenesch F, Etienne J, Freney J. 2002. Population diversity of Staphylococcus intermedius isolates from various host species: Typing by 16S-23S intergenic ribosomal DNA spacer polymorphism analysis. Journal of Clinical Microbiology. 40(6):2275-7. https://doi.org/10.1128/JCM.40.6.2275-2277.2002
Black CC, Solyman SM, Eberlein LC, Bemis DA, Woron AM, Kania SA. 2009. Identification of a predominant multilocus sequence type, pulsed-field gel electrophoresis cluster, and novel staphylococcal chromosomal cassette in clinical isolates of mecA-containing, methicillin-resistant Staphylococcus pseudintermedius. Veterinary Microbiology. 139(3-4):333-8. https://doi.org/10.1016/j.vetmic.2009.06.029
Blaiotta G, Fusco V, Ercolini D, Pepe O, Coppola S. 2010. Diversity of Staphylococcus species strains based on partial kat (catalase) gene sequences and design of a PCR-Restriction Fragment Length Polymorphism assay for identification and differentiation of coagulase-positive species (S. aureus, S. delphini, S. hyicus, S. intermedius, S. pseudintermedius, and S. schleiferi subsp. coagulans). Journal of Clinical Microbiology. 48(1):192-201. https://doi.org/10.1128/JCM.00542-09
Bobenchik A, Charnot-Katsikas A, Westblade L. 2017. What does the CLSI AST Subcommittee do?. CLSI AST News Update. 2(1):1-17
Börjesson S, Gómez-Sanz E, Ekström K, Torres C, Grönlund U. 2015. Staphylococcus pseudintermedius can be misdiagnosed as Staphylococcus aureus in humans with dog bite wounds. European Journal of Clinical Microbiology and Infectious Diseases. 34(4):839-44. https://doi.org/10.1007/s10096-014-2300-y
Campanile F, Bongiorno D, Borbone S, Venditti M, Giannella M, Franchi C, Stefani S. 2007. Characterization of a variant of the SCCmec element in a bloodstream isolate of Staphylococcus intermedius. Microbial Drug Resistance. 13(1):7-10. https://doi.org/10.1089/mdr.2006.9991
Cheung GY, Joo HS, Chatterjee SS, Otto M. 2014. Phenol-soluble modulins - critical determinants of staphylococcal virulence. FEMS Microbiology Reviews. 38(4):698-719. https://doi.org/10.1111/1574-6976.12057
Chrobak-Chmiel D, Golke A, Dembele K, Ćwiek K, Kizerwetter-Świda M, Rzewuska M, Binek M. 2018. Staphylococcus pseudintermedius, both commensal and pathogen. Medycyna Weterynaryjna. 74(6):362-70. https://doi.org/10.21521/mw.6042
Chrobak D, Kizerwetter-Świda M, Rzewuska M, Moodley A, Guardabassi L, Binek M. 2011. Molecular characterization of Staphylococcus pseudintermedius strains isolated from clinical samples of animal origin. Folia Microbiologica. 56(5):415-22. https://doi.org/10.1007/s12223-011-0064-7
Corrò M, Skarin J, Börjesson S, Rota A. 2018. Occurrence and characterization of methicillin-resistant Staphylococcus pseudintermedius in successive parturitions of bitches and their puppies in two kennels in Italy. BMC Veterinary Research. 14(1):1-8. https://doi.org/10.1186/s12917-018-1612-z
Couto N, Belas A, Oliveira M, Almeida P, Clemente C, Pomba C. 2015. Comparative RNA-seq-based transcriptome analysis of the virulence characteristics of methicillin-resistant and -susceptible Staphylococcus pseudintermedius strains isolated from small animals. Antimicrobial Agents and Chemotherapy. 60(2):962-7. https://doi.org/10.1128/AAC.01907-15
Davis MF, Iverson SA, Baron P, Vasse A, Silbergeld EK, Lautenbach E, Morris DO. 2012. Household transmission of methicillin-resistant Staphylococcus aureus and other staphylococci. The Lancet Infectious Diseases. 12(9):703-16. https://doi.org/10.1016/S1473-3099(12)70156-1
Decristophoris P, Fasola A, Benagli C, Tonolla M, Petrini O. 2011. Identification of Staphylococcus intermedius Group by MALDI-TOF MS. Systematic and Applied Microbiology. 34(1):45-51. https://doi.org/10.1016/j.syapm.2010.11.004
Descloux S, Rossano A, Perreten V. 2008. Characterization of new staphylococcal cassette chromosome mec (SCCmec) and topoisomerase genes in fluoroquinolone- and methicillin-resistant Staphylococcus pseudintermedius. Journal of Clinical Microbiology. 46(5):1818-23. https://doi.org/10.1128/JCM.02255-07
Devriese LA, Nzuambe D, Godard C. 1985. Identification and characteristics of staphylococci isolated from lesions and normal skin of horses. Veterinary Microbiology. 10(3):269-77. https://doi.org/10.1016/0378-1135(85)90052-5
Devriese LA, Vancanneyt M, Baele M, Vaneechoutte M, De Graef E, Snauwaert C, Cleenwerck I, Dawyndt P, Swings J, Decostere A, Haesebrouck FC. 2005. Staphylococcus pseudintermedius sp. nov., a coagulase-positive species from animals. International Journal of Systematic and Evolutionary Microbiology. 55(4):1569-73. https://doi.org/10.1099/ijs.0.63413-0
Edwards VM, Deringer JR, Callantine SD, Deobald CF, Berger PH, Kapur V, Stauffacher CV, Bohach GA. 1997. Characterization of the canine type c enterotoxin produced by Staphylococcus intermedius pyoderma isolates. Infection and Immunity. 65(6):2346-52. https://doi.org/10.1128/iai.65.6.2346-2352.1997
Federación de Asociaciones Veterinarias de Animales de Compañía Europeas (FECAVA). 2018. Recomendaciones FECAVA para el tratamiento adecuado con antibióticos. [En línea] disponible en https://fecava.org/wp-content/uploads/2020/01/FECAVA-Recommendations-for-Appropriate-Antimicrobial-SPANISH.pdf. [Consultado 20/08/2020].
Fazakerley J, Nuttall T, Sales D, Schmidt V, Carter SD, Hart CA, McEwan NA. 2009. Staphylococcal colonization of mucosal and lesional skin sites in atopic and healthy dogs. Veterinary Dermatology. 20(3):179-84. https://doi.org/10.1111/j.1365-3164.2009.00745.x
Ference EH, Danielian A, Kim HW, Yoo F, Kuan EC, Suh JD. 2019. Zoonotic Staphylococcus pseudintermedius sinonasal infections: risk factors and resistance patterns. International Forum of Allergy and Rhinology. 9(7):724-9. https://doi.org/10.1002/alr.22329
Foster TJ. 2017. Antibiotic resistance in Staphylococcus aureus. Current status and future prospects. FEMS Microbiology Reviews. 41:430-49. https://doi.org/10.1093/femsre/fux007
Futagawa-Saito K, Makino S, Sunaga F, Kato Y, Sakurai-Komada N, Ba-Thein W, Fukuyasu T. 2009. Identification of first exfoliative toxin in Staphylococcus pseudintermedius. FEMS Microbiology Letters. 301(2):176-80. https://doi.org/10.1111/j.1574-6968.2009.01823.x
Futagawa-Saito K, Sugiyama T, Karube S, Sakurai N, Ba-Thein W, Fukuyasu T. 2004a. Prevalence and characterization of leukotoxin-producing Staphylococcus intermedius in isolates from dogs and pigeons. Journal of Clinical Microbiology. 42(11):5324-26. https://doi.org/10.1128/JCM.42.11.5324-5326.2004
Futagawa-Saito K, Suzuki M, Ohsawa M, Ohshima S, Sakurai N, Ba-Thein W, Fukuyasu T. 2004b. Identification and prevalence of an enterotoxin-ralated gene, se-int, in Staphylococcus intermedius isolates from dogs and pigeons. Journal of Applied Microbiology. 96(6):1361-6. https://doi.org/10.1111/j.1365-2672.2004.02264.x
Gagetti P, Errecalde L, Wattam AR, De Belder D, Ojeda Saavedra M, Corso A, Rosato AE. 2020. Characterization of the first mecA-positive multidrug-resistant Staphylococcus pseudintermedius isolated from an Argentinian patient. Microbial Drug Resistance. 26(7):717-21. https://doi.org/10.1089/mdr.2019.0308
Gagetti P, Wattam AR, Giacoboni G, De Paulis A, Bertona E, Corso A, Rosato AE. 2019. Identification and molecular epidemiology of methicillin resistant Staphylococcus pseudintermedius strains isolated from canine clinical samples in Argentina. BMC Veterinary Research.15(1):264. https://doi.org/10.1186/s12917-019-1990-x
Gavidia Catalán V, Talavera M. 2012. La construcción del concepto de salud. Didáctica de las Ciencias Experimentales y Sociales. 26:161-75. https://doi.org/10.7203/dces.26.1935
Geoghegan JA, Smith EJ, Speziale P, Foster TJ. 2009. Staphylococcus pseudintermedius expresses surface proteins that closely resemble those from Staphylococcus aureus. Veterinary Microbiology.138(3-4):345-52. https://doi.org/10.1016/j.vetmic.2009.03.030
Geraghty L, Booth M, Rowan N, Fogarty A. 2013. Investigations on the efficacy of routinely used phenotypic methods compared to genotypic approaches for the identification of staphylococcal species isolated from companion animals in Irish veterinary hospitals. Irish Veterinary Journal. 66(1):1-9. https://doi.org/10.1186/2046-0481-66-7
Gharsa H, Ben Slama K, Gómez-Sanz E, Lozano C, Klibi N, Jouini A, Messadi L, Boudabous A, Torres C. 2013. Antimicrobial resistance, virulence genes, and genetic lineages of Staphylococcus pseudintermedius in healthy dogs in Tunisia. Microbial Ecology. 66(2):363-8. https://doi.org/10.1007/s00248-013-0243-y
Gold RM, Cohen ND, Lawhon SD. 2014. Amikacin resistance in Staphylococcus pseudintermedius isolated from dogs. Journal of Clinical Microbiology. 52(10):3641-6. https://doi.org/10.1128/JCM.01253-14
Gómez-Sanz E, Torres C, Lozano C, Sáenz Y, Zarazaga M. 2011. Detection and characterization of methicillin-resistant Staphylococcus pseudintermedius in healthy dogs in La Rioja, Spain. Comparative Immunology, Microbiology and Infectious Diseases. 34(5):447-53. https://doi.org/10.1016/j.cimid.2011.08.002
Gómez-Sanz E, Torres C, Lozano C, Zarazaga M. 2013. High diversity of Staphylococcus aureus and Staphylococcus pseudintermedius lineages and toxigenic traits in healthy pet-owning household members. Underestimating normal household contact?’. Comparative Immunology, Microbiology and Infectious Diseases. 36(1):83-94. https://doi.org/10.1016/j.cimid.2012.10.001
Gortazar C, Reperant LA, Kuiken T, de la Fuente J, Boadella M, Martínez-Lopez B, Ruiz-Fons F, Estrada-Peña A, Drosten C, Medley G, Ostfeld R, Peterson T, VerCauteren KC, Menge C, Artois M, Schultsz C, Delahay R, Serra-Cobo J, Poulin R, Keck F, Aguirre AA, Henttonen H, Dobson AP, Kutz S, Lubroth J, Mysterud A. 2014. Crossing the interspecies barrier: opening the door to zoonotic pathogens. PLoS Pathogens. 10(6): e1004129. https://doi.org/10.1371/journal.ppat.1004129
Gortel K, Campbell KL, Kakoma I, Whittem T, Schaeffer DJ, Weisiger RM. 1999. Methicillin resistance among staphylococci isolated from dogs. American Journal of Veterinary Research. 60(12):1526-30.
Grandolfo, E. 2018. Looking through Staphylococcus pseudintermedius infections: Could Spa be considered a possible vaccine target?. Virulence. 9(1):703-6. https://doi.org/10.1080/21505594.2018.1426964
Guardabassi L, Damborg P, Stamm I, Kopp PA, Broens EM, Toutain PL. 2017. Diagnostic microbiology in veterinary dermatology: present and future. Veterinary Dermatology. 28(1):146-e30. https://doi.org/10.1111/vde.12414
Guardabassi L, Schwarz S, Lloyd DH. 2004. Pet animals as reservoirs of antimicrobial-resistant bacteria. Journal of Antimicrobial Chemotherapy. 54(2):321-2. https://doi.org/10.1093/jac/dkh332
Hajek V. 1976. Staphylococcus intermedius, a new species isolated from animals. International Journal of Systematic and Evolutionary Microbiology. 26:401-8. https://doi.org/10.1099/00207713-26-4-401.
Hartmann FA, White DG, West SE, Walker RD, Deboer DJ. 2005. Molecular characterization of Staphylococcus intermedius carriage by healthy dogs and comparison of antimicrobial susceptibility patterns to isolates from dogs with pyoderma. Veterinary Microbiology. 108(1-2):119-31. https://doi.org/10.1016/j.vetmic.2005.03.006
Hesselbarth J, Flachsbarth MF, Amtsberg G. 1994. Studies on the production of an exfoliative toxin by Staphylococcus intermedius. Journal of Veterinary Medicine, Series B, 41:411-16. https://doi.org/10.1111/j.1439-0450.1994.tb00245.x
Hesselbarth J, Schwarz S. 1995. Comparative ribotyping of Staphylococcus intermedius from dogs, pigeons, horses and mink. Veterinary Microbiology. 45(1):11-7. https://doi.org/10.1016/0378-1135(94)00125-G
Iyori K, Hisatsune J, Kawakami T, Shibata S, Murayama N, Ide K, Nagata M, Fukata T, Iwasaki T, Oshima K, Hattori M, Sugai M, Nishifuji K. 2010. Identification of a novel Staphylococcus pseudintermedius exfoliative toxin gene and its prevalence in isolates from canines with pyoderma and healthy dogs. FEMS Microbiology Letters. 312(2):169-75. https://doi.org/10.1111/j.1574-6968.2010.02113.x
Kadlec K, Schwarz S. 2012. Antimicrobial resistance of Staphylococcus pseudintermedius. Veterinary Dermatology. 23(4):19-25. https://doi.org/10.1111/j.1365-3164.2012.01056.x
Kadlec K, Schwarz S, Perreten V, Andersson UG, Finn M, Greko C, Moodley A, Kania SA, Frank LA, Bemis DA, Franco A, Iurescia M, Battisti A, Duim B, Wagenaar JA, van Duijkeren E, Weese JS, Fitzgerald JR, Rossano A, Guardabassi L. 2010. Molecular analysis of methicillin-resistant Staphylococcus pseudintermedius of feline origin from different European countries and North America. Journal of Antimicrobial Chemotherapy. 65:1826-8. https://doi.org/10.1093/jac/dkq203
Kadlec K, van Duijkeren E, Wagenaar JA, Schwarz S. 2011. Molecular basis of rifampicin resistance in methicillin-resistant Staphylococcus pseudintermedius isolates from dogs. Journal of Antimicrobial Chemotherapy. 66(6):1236-42. https://doi.org/10.1093/jac/dkr118
Kawano J, Shimizu A, Kimura S. 1982. Isolation of bacteriophages for typing Staphylococcus intermedius isolated from pigeons. Zentralblatt fur Bakteriologie Mikrobiologie und Hygiene - Abt. 1 Orig. A, 251(4):487-93. https://doi.org/10.1016/s0174-3031(82)80131-5
Kempker R, Mangalat D, Kongphet-Tran T, Eaton M. 2009. Beware of the pet dog: A case of Staphylococcus intermedius infection. American Journal of the Medical Sciences. 338(5):425-7. https://doi.org/10.1097/MAJ.0b013e3181b0baa9
Kmieciak. W, Szewczyk EM. 2018. Are zoonotic Staphylococcus pseudintermedius strains a growing threat for humans?. Folia Microbiologica. 63(6):743-7. https://doi.org/10.1007/s12223-018-0615-2
Lakhundi S, Zhang K. 2018. Methicillin-resistant Staphylococcus aureus: molecular characterization, evolution, and epidemiology. Clinical Microbiology Reviews. 31:e00020-18. https://doi.org/10.1128/CMR.00020-18
Lautz S, Kanbar T, Alber J, Lämmler C, Weiss R, Prenger-Berninghoff E, Zschöck M. 2006. Dissemination of the gene encoding exfoliative toxin of Staphylococcus intermedius among strains isolated from dogs during routine microbiological diagnostics. Journal of Veterinary Medicine Series B: Infectious Diseases and Veterinary Public Health, 53(9):434-8. https://doi.org/10.1111/j.1439-0450.2006.00999.x
Lilenbaum W, Esteves AL, Souza GN. 1998. Prevalence and antimicrobial susceptibility of staphylococci isolated from saliva of clinically normal cats. Letters in Applied Microbiology. 28(6):448-52. https://doi.org/10.1046/j.1365-2672.1999.00540.x
Linares-Rodríguez JF, Martínez-Menéndez JL. 2005. Antimicrobial resistance and bacterial virulence. Enfermedades Infecciosas y Microbiología Clínica. 23(2):86-93. https://doi.org/10.1157/13071612
Loeffler A, Linek M, Moodley A, Guardabassi L, Sung JML, Winkler M, Weiss R, Lloyd DH. 2007. First report of multiresistant, mecA-positive Staphylococcus intermedius in Europe: 12 cases from a veterinary dermatology referral clinic in Germany. Veterinary Dermatology.18(6):412-21. https://doi.org/10.1111/j.1365-3164.2007.00635.x
Loeffler A, Lloyd DH. 2018. What has changed in canine pyoderma? A narrative review. Veterinary Journal. 235:73-82. https://doiorg/10.1016/j.tvjl.2018.04.002
Lozano C, Rezusta A, Ferrer I, Pérez-Laguna V, Zarazaga M, Ruiz-Ripa L, Revillo MJ, Torres C. 2017. Staphylococcus pseudintermedius human infection cases in Spain: dog-to-human transmission. Vector-Borne and Zoonotic Diseases. 17(4):268-70. https://doi.org/10.1089/vbz.2016.2048
Lueddeke GR, Kaufman GE, Kahn LH, Krecek RC, Willingham AL, Stroud CM, Lindenmayer JM, Kaplan B, Conti LA, Monath TP, Woodall J. 2016. Preparing society to create the world we need through ‘One Health’ education, South Eastern European Journal of Public Health (SEEJPH). https://doi.org/10.4119/seejph-1841.
Maali Y, Martins-Simões P, Valour F, Bouvard D, Rasigade J-P, Bes M, Haenni M, Ferry T, Laurent F, Trouillet-Assant S. 2016. Pathophysiological mechanisms of Staphylococcus non-aureus bone and joint infection: Interspecies homogeneity and specific behavior of S. pseudintermedius. Frontiers in Microbiology. 7:1-9. https://doi.org/10.3389/fmicb.2016.01063.
Magiorakos AP, Srinivasan A, Carey RB, Carmeli Y, Falagas ME, Giske CG, Harbarth S, Hindler JF, Kahlmeter G, Olsson-Liljequist B, Paterson DL, Rice LB, Stelling J, Struelens MJ, Vatopoulos A, Weber JT, Monnet DL. 2011. Multidrug-resistant, extensively drug-resistant and pandrug-resistant bacteria: an international expert proposal for interim standard definitions for acquired resistance. Clinical Microbiology and Infection. 18(3):268-81. https://doi.org/10.1111/j.1469-0691.2011.03570.x
McCune S, McCardle P, Griffin J, Esposito L, Hurley K, Bures R, Kruger KA. 2020. Editorial: Human-animal interaction (HAI) research: A decade of progress. Frontiers in Veterinary Science. 7:44. https://doi.org/10.3389/fvets.2020.00044
McEwan NA, Mellor D, Kalna G. 2006. Adherence by Staphylococcus intermedius to canine corneocytes: A preliminary study comparing noninflamed and inflamed atopic canine skin. Veterinary Dermatology. 17(2):151-4. https://doi.org/10.1111/j.1365-3164.2006.00503.x.
Momota Y, Shimada K, Noguchi A, Saito A, Nozawa S, Niina A, Tani K, Azakami D, Ishioka K, Sako T. 2016. The modified corneocyte surface area measurement as an index of epidermal barrier properties: Inverse correlation with transepidermal water loss. Veterinary Dermatology. 27(2):67-e19. https://doi.org/10.1111/vde.12287
Moodley A, Damborg P, Nielsen SS. 2014. Antimicrobial resistance in methicillin susceptible and methicillin resistant Staphylococcus pseudintermedius of canine origin: Literature review from 1980 to 2013. Veterinary Microbiology. 171(3-4):337-41. https://doi.org/10.1016/j.vetmic.2014.02.008
Moreira CA, de Oliveira LC, Silveira Mendes M, de Melo Santiago T, Bedê Barros E, Barreto Mano de Carvalho C. 2012. Biofilm production by clinical staphylococci strains from canine otitis. Brazilian Journal of Microbiology. 43(1):371-4. https://doi.org/10.1590/S1517-83822012000100044
Murray AK, Lee J, Bendall R, Zhang L, Sunde M, Schau Slettemeas J, Gaze W, Page AJ, Vos M. 2018. Staphylococcus cornubiensis sp. nov., a member of the Staphylococcus intermedius group (SIG). International Journal of Systematic and Evolutionary Microbiology. 68(11):3404-8. https://doi.org/10.1099/ijsem.0.002992
Norström M, Sunde M, Tharaldsen H, Mørk T, Bergsjø B, Kruse H. 2009. Antimicrobial resistance in Staphylococcus pseudintermedius in the Norwegian dog population. Microbial Drug Resistance. 15(1):55-9. https://doi.org/10.1089/mdr.2009.0865
Organización Mundial de Sanidad Animal (OIE). 2020. Una sola salud [En línea] Disponible en: https://oie.int/es/para-los-periodistas/una-sola-salud/ [Consultado 20/08/2020].
Organización Mundial de Sanidad Animal (OIE) 2010. The FAO-OIE-WHO Collaboration. A Tripartite Concept Note. [En línea] Disponible en: https://oie.int/fileadmin/Home/eng/Current_Scientific_Issues/docs/pdf/FINAL_CONCEPT_NOTE_Hanoi.pdf [Consultado 20/08/2020].
Osland AM, Vestby LK, Fanuelsen H, Slettemeås JS, Sunde M. 2012. Clonal diversity and biofilm-forming ability of methicillin-resistant Staphylococcus pseudintermedius. Journal of Antimicrobial Chemotherapy. 67(4):841-8. https://doi.org/10.1093/jac/dkr576
Pellerin JL, Bourdeau P, Sebbag H, Person JM. 1998. Epidemiosurveillance of antimicrobial compound resistance of Staphylococcus intermedius clinical isolates from canine pyodermas. Comparative Immunology, Microbiology and Infectious Diseases. 21(2):115-33. https://doi.org/10.1016/S0147-9571(97)00026-X
Perreten V, Chanchaithong P, Prapasarakul N, Rossano A, Blum SE, Elad D, Schwendener S. 2013. Novel pseudo-staphylococcal cassette chromosome mec element (ΨSCCmec57395) in methicillin-resistant Staphylococcus pseudintermedius CC45. Antimicrobial Agents and Chemotherapy, 57(11):5509-15. https://doi.org/10.1128/AAC.00738-13
Perreten V, Kadlec K, Schwarz S, Gronlund Andersson U, Finn M, Greko C, Moodley A, Kania SA, Frank LA, Bemis DA, Franco A, Iurescia M, Battisti A, Duim B, Wagenaar JA, van Duijkeren E, Weese JS, Fitzgerald JR, Rossano A, Guardabassi L. 2010. Clonal spread of methicillin-resistant Staphylococcus pseudintermedius in Europe and North America: an international multicentre study. Journal of Antimicrobial Chemotherapy. 65:1145-54. https://doi.org/10.1093/jac/dkq078
Perreten V, Kania S, Bemis D. 2020. Staphylococcus ursi sp. nov., a new member of the “Staphylococcus intermedius group” isolated from healthy black bears. International Journal of Systematic and Evolutionary Microbiology. 10973936:1-9. https://doi.org/10.1099/ijsem.0.004324
Pomba C, Rantala M, Greko C, BaptisteCKE, Catry B, van Duijkeren E, Mateus A, Moreno MA, Pyorala S , Ruzauskas M, Sanders P, Teale C, Threlfall EJ, Kunsagi Z, Torren-Edo J, Jukes H and Torneke K. 2017. Public health risk of antimicrobial resistance transfer from companion animals. Journal of Antimicrobial Chemotherapy. 72(4):957-68. https://doi.org/10.1093/jac/dkw481
Ross Fitzgerald J. 2009. The Staphylococcus intermedius group of bacterial pathogens: Species re-classification, pathogenesis and the emergence of methicillin resistance. Veterinary Dermatology. 20(5–6):490-5. https://doi.org/10.1111/j.1365-3164.2009.00828.x
Santos FCO, de Souza MV, Lühers Graça D, Vargas A, do Carmo Lopes Moreira J, Mota Zandim B. 2008. Piodermite profunda por Staphylococcus intermedius em eqüino. Ciencia Rural 38(9):2641-5. https://doi.org/10.1590/S0103-84782008000900040
Sasaki T, Kikuchi K, Tanaka Y, Takahashi N, Kamata S, Hiramatsu K. 2007. Reclassification of phenotypically identified Staphylococcus intermedius strains. Journal of Clinical Microbiology. 45(9):2770-8. https://doi.org/10.1128/JCM.00360-07.
Sasaki T, Tsubakishita S, Tanaka Y, Sakusabe A, Ohtsuka M, Hirotaki S, Kawakami T, Fukata T, Hiramatsu K. 2010. Multiplex-PCR method for species identification of coagulase-positive staphylococci. Journal of Clinical Microbiology. 48(3):765-9. https://doi.org/10.1128/JCM.01232-09
Savini V, Barbarini D, Polakowska K, Gherardi G, Białecka A, Kasprowicz A, Polilli E, Marrollo R, Di Bonaventura G, Fazii P, D'Antonio D, Miedzobrodzki J, Carretto E. 2013. Methicillin-resistant Staphylococcus pseudintermedius infection in a bone marrow transplant recipient. Journal of Clinical Microbiology. 51(5):1636-8. https://doi.org/10.1128/JCM.03310-12
Schleifer KH, Bell JA. 2010. LPSN - List of Prokaryotic names with Standing in Nomenclature. Género Staphylococcus [En línea] disponible en https://lpsn.dsmz.de/genus/staphylococcus [Consultado 20/08/2020].
Simou C, Thoday KL, Forsythe PJ, Hill PB. 2005. Adherence of Staphylococcus intermedius to corneocytes of healthy and atopic dogs: Effect of pyoderma, pruritus score, treatment and gender. Veterinary Dermatology. 16(6):385-91. https://doi.org/10.1111/j.1365-3164.2005.00484.x
Singh A, Walker M, Rosseau J, Weese SJ. 2013. Characterization of the biofilm forming ability of Staphylococcus pseudintermedius from dogs. BMC Veterinary Research, 9:93. https://doi.org/10.1186/1746-6148-9-93.
Somayaji R, Priyantha MAR, Rubin JE, Church D. 2016. Human infections due to Staphylococcus pseudintermedius, an emerging zoonosis of canine origin: report of 24 cases. Diagnostic Microbiology and Infectious Diseases. 85(4):471-6. https://doi.org/10.1016/j.diagmicrobio.2016.05.008
Stegmann R, Burnens A, Maranta CA, Perreten V. 2010. Human infection associated with methicillin-resistant Staphylococcus pseudintermedius ST71. Journal of Antimicrobial Chemotherapy. 65(9):2047-8. https://doi.org/10.1093/jac/dkq241
SWEDRES-SVARM 2015. Use of Antimicrobials and Occurrence of Antimicrobial Resistance in Sweden. Solna/Uppsala, Sweden. [En línea] disponible en https://folkhalsomyndigheten.se/contentassets/e52354e8f91b43b9b25186f06b7a1b48/swedres-svarm-2015-15099.pdf. [Consultado 20/08/2020].
Talan DA, Staatz D, Staatz A, Goldstein EJ, Singer K, Overturf GD. 1989. Staphylococcus intermedius in canine gingiva and canine-inflicted human wound infections: Laboratory characterization of a newly recognized zoonotic pathogen. Journal of Clinical Microbiology. 27(1):78-81. https://doi.org/10.1128/jcm.27.1.78-81.1989
Terauchi R, Sato H, Hasegawa T, Yamaguchi T, Aizawa C, Maehara N. 2003. Isolation of exfoliative toxin from Staphylococcus intermedius and its local toxicity in dogs. Veterinary Microbiology. 94(1):19-29. https://doi.org/10.1016/S0378-1135(03)00048-8
Vallat B. 2009. Un mundo, una salud. OIE Boletín No 2009-2, 2:2-9. [En línea] disponible en https://oie.int/fileadmin/Home/esp/Publications_%26_Documentation/docs/pdf/bulletin/Bull_2009-2-ESP.pdf [Consultado 20/08/2020].
Vandenesch F, Celard M, Arpin D, Bes M, Greenland T, Etienne J. 1995. Catheter-related bacteremia associated with coagulase-positive Staphylococcus intermedius. Journal of Clinical Microbiology. 33(9):2508-10. https://doi.org/10.1128/jcm.33.9.2508-2510.1995
van Duijkeren E, Houwers DJ, Schoormans A, Broekhuizen-Stins MJ. 2008. Transmission of methicillin-resistant Staphylococcus intermedius between humans and animals. Veterinary Microbiology. 128(1–2):213-5. https://doi.org/10.1016/j.vetmic.2007.11.016
van Duijkeren E, Catry B, Greko C, Moreno MA, Pomba MC, Pyörälä S, Ruzauskas M, Sanders P, Threlfall EJ, Torren-Edo J, Törneke K. 2011. Review on methicillin-resistant Staphylococcus pseudintermedius. Journal of Antimicrobial Chemotherapy. 66(12):2705-14. https://doi.org/10.1093/jac/dkr367
van Hoovels L, Vankeerberghen A, Boel A, van Vaerenbergh K, De Beenhouwer H. 2006. First case of Staphylococcus pseudintermedius infection in a human. Journal of Clinical Microbiology. 44(12):4609-12. https://doi.org/10.1128/JCM.01308-06
Verstappen KM, Huijbregts L, Spaninks M, Wagenaar JA, Fluit AC, Duim B. 2017. Development of a real-time PCR for detection of Staphylococcus pseudintermedius using a novel automated comparison of whole-genome sequences. PLoS ONE. 12(8): e0183925. https://doi.org/10.1371/journal.pone.0183925
Vigo GB, Giacoboni GI, Gagetti PS, Pasterán FG, Corso AC. 2015. Resistencia antimicrobiana y epidemiología molecular de aislamientos de Staphylococcus pseudintermedius de muestras clínicas de caninos, Revista Argentina de Microbiologia. 47(3):206-11. https://doi.org/10.1016/j.ram.2015.06.002
Walther B, Hermes J, Cuny C, Wieler HL, Vincze S, Elnaga YA, Stamm I, Kopp PA, Kohn B, Witte W, Jansen A, Conraths FJ, Semmler T, Eckmanns T, Lubke-Becker A. 2012. Sharing more than friendship - nasal colonization with coagulase-positive staphylococci (CPS) and co-habitation aspects of dogs and their owners. PLoS ONE. 7(4): e35197. https://doi.org/10.1371/journal.pone.0035197.
Weese JS. 2012. Staphylococcal control in the veterinary hospital. Veterinary Dermatology. 23(4): 292-e58. https://doi.org/10.1111/j.1365-3164.2012.01048.x
Weese JS, van Duijkeren E. 2010. Methicillin-resistant Staphylococcus aureus and Staphylococcus pseudintermedius in veterinary medicine. Veterinary Microbiology. 140(3-4):418-29. https://doi.org/10.1016/j.vetmic.2009.01.039
Wettstein K, Descloux S, Rossano A, Perreten V. 2008. Emergence of methicillin-resistant Staphylococcus pseudintermedius in Switzerland: three cases of urinary tract infections in cats. Schweizer Archiv für Tierheilkunde. 150(7):339-43. https://doi.org/10.1024/0036-7281.150.7.339
Williams CJ, Scheftel JM, Elchos BL, Hopkins SG, Levine JF. 2015. Compendium of veterinary standard precautions for zoonotic disease prevention in veterinary personnel: National Association of State Public Health Veterinarians: Veterinary Infection Control Committee. Journal of the American Veterinary Medical Association. 247(11):1252-77. [Correcciones publicadas en Journal of American Veterinary Medical Association. 2016.15;248(2):171]. https://doi.org/10.2460/javma.247.11.1252
Woolley KL, Kelly RF, Fazakerley J, Williams NJ, Nuttall TJ, McEwan NA. 2008. Reduced in vitro adherence of Staphylococcus species to feline corneocytes compared to canine and human corneocytes. Veterinary Dermatology. 19(1):1-6. https://doi.org/10.1111/j.1365-3164.2007.00649.x
Yoon JW, Lee GJ, Lee SY, Park C, Yoo JH, Park HM. 2010. Prevalence of genes for enterotoxins, toxic shock syndrome toxin 1 and exfoliative toxin among clinical isolates of Staphylococcus pseudintermedius from canine origin. Veterinary Dermatology. 21(5):484-9. https://doi.org/10.1111/j.1365-3164.2009.00874.x