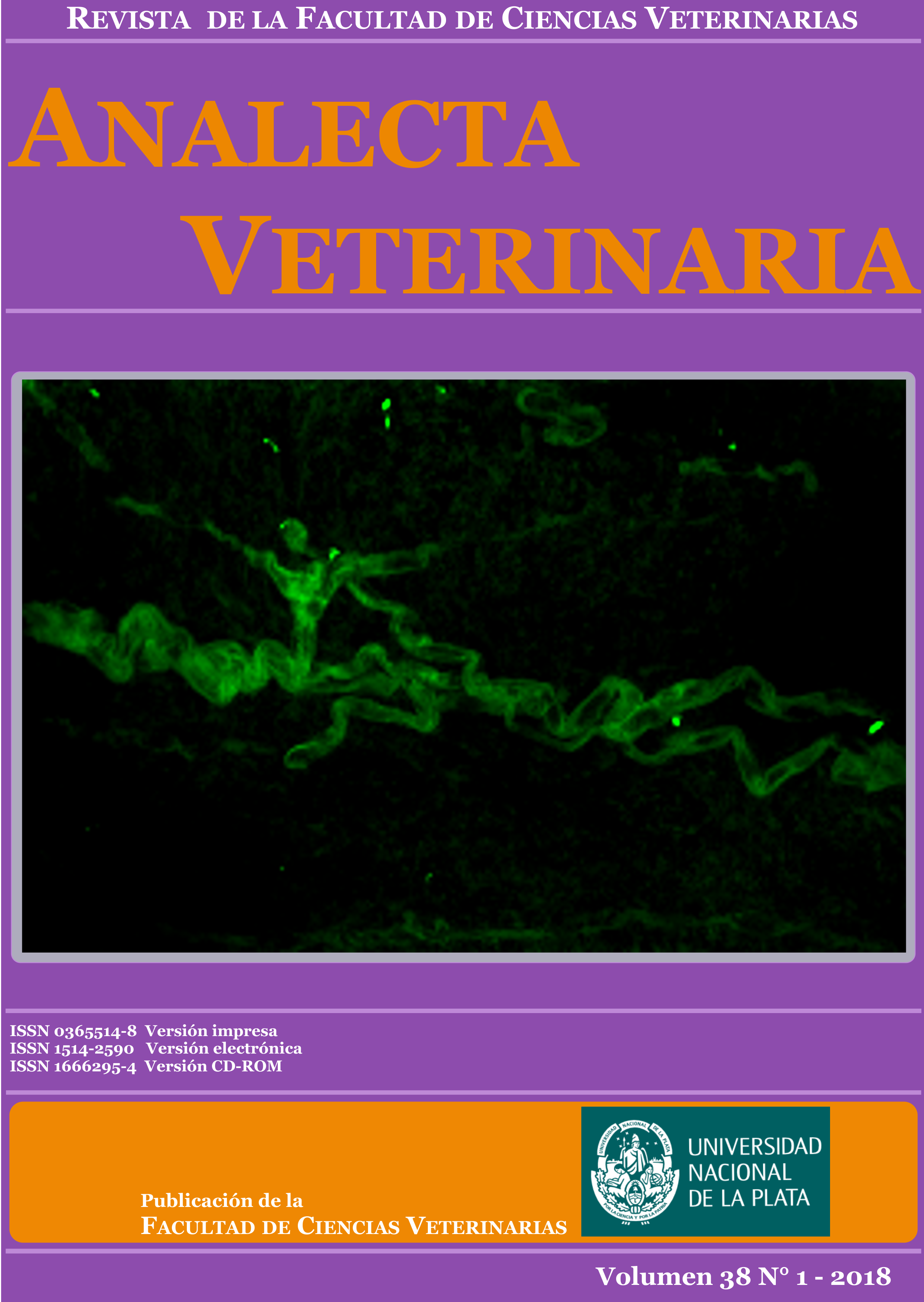
Uso de la técnica CLARITY para la identificación de marcadores fluorescentes en cortes gruesos de la médula espinal
DOI:
https://doi.org/10.24215/15142590e021Palabras clave:
Fluorescencia, microscopía confocal, procesamientoResumen
El estudio de las interconexiones que establecen las neuronas entre sí o con las células de la glía es uno de los mayores desafíos de la neurociencia. Los anticuerpos fluorescentes permiten la identificación y localización de diferentes estructuras tisulares. Sin embargo, su uso eficiente requiere de la utilización de cortes delgados de muestras, que constituyen una limitación para el estudio de las interconexiones en el sistema nervioso. Asimismo, un mayor espesor de la muestra limita la penetración de la luz emitida por el microscopio, mientras que la opacidad propia del tejido nervioso debida a su alto contenido lipídico dificulta la adquisición de las imágenes al restringir la resolución de los objetos. La técnica CLARITY (del inglés, Clear, Lipidexchanged, Acrylamidehybridized Rigid, Imaging/Immunostaining/In situ hybridizationcompatible, Tissue hYdrogel) permite subsanar estos inconvenientes. Esta técnica fue adaptada en nuestro laboratorio para el estudio de la médula espinal de rata. De acuerdo con el procedimiento realizado, pudimos obtener un órgano completamente translúcido, estructuralmente intacto y que permitió su procesamiento mediante técnicas de inmunofluorescencia y de lectinhistoquímica sin mayores dificultades. A partir de muestras de más de 1 mm de espesor se obtuvieron imágenes confocales de gran resolución y de mayor penetrabilidad que las que se obtienen utilizando las técnicas de procesamiento convencionales. La implementación de esta técnica en nuestro laboratorio permitirá optimizar la información obtenida a partir de muestras de interés.
Referencias
Chung K y Deisseroth K. 2013. CLARITY for mapping the nervous system. Nature Methods. 2013 Jun;10(6):50813. doi: 10.1038/nmeth.2481
Ertürk A y Bradke F. 2013. Highresolution imaging of entire organs by 3dimensional imaging of solvent cleared organs (3DISCO). Experimental Neurology. 242:5764. doi: 10.1016/j.expneurol.2012.10.018
Grabinski TM, Kneynsberg A, Manfredsson FP, Kanaan NM. 2015. A method for combining RNAscope in situ hybridization with immunohistochemistry in thick freefloating brain sections and primary neuronal cultures. PLoS One. 10(3):e0120120. doi: 10.1371/journal.pone.0120120
Ke MT, Fujimoto S, Imai T. 2013. SeeDB: a simple and morphologypreserving optical clearing agent for neuronal circuit reconstruction. Nature Neuroscience. 6(8):115461. doi: 10.1038/nn.3447
Kim SY, Chung K, Deisseroth K. 2013. Light microscopy mapping of connections in the intact rain. Trends Cognitive Sciences. 17(12):5969. doi: 10.1016/j.tics.2013.10.005
Kuwajima T, Sitko AA, Bhansali P, Jurgens C, Guido W, Mason C. 2013. ClearT: a detergent and solventfree clearing method for neuronal and nonneuronal tissue. Development. 140(6):13648. doi: 10.1242/dev.091844
Lee H, Park JH, Seo I, Park SH, Kim S. 2014. Improved application of the electrophoretic tissue clearing technology, CLARITY, to intact solid organs including brain, pancreas, liver, kidney, lung, and intestine. BMC Developmental Biology. 21;14:48. doi: 10.1186/s1286101400483
Orlich M, Kiefer F. 2017. A qualitative comparison of ten tissue clearing techniques. Histology & Histopathology. 12:11903. doi: 10.14670/HH11903
Portiansky EL. 2013. Análisis multidimensional de imágenes digitales. La Plata, Argentina. Universidad Nacional de La Plata, http://hdl.handle.net/10915/48218.
Tomer R, Ye L, Hsueh B, Deisseroth K. 2014. Advanced CLARITY for rapid and highresolution imaging of intact tissues. Nature Protocols. 9(7): 168297. doi: 10.1038/nprot.2014.123
Zheng H y Rinaman L. 2015. Simplified CLARITY for visualizing immunofluorescence labelling in the developing rat brain. Brain Structure & Function. 221(4):237583. doi: 10.1007/s0042901510200
Descargas
Publicado
Número
Sección
Licencia
Los autores/as conservan los derechos de autor y ceden a la revista el derecho de la primera publicación, con el trabajo registrado con la licencia de atribución de Creative Commons, que permite a terceros utilizar lo publicado siempre que mencionen la autoría del trabajo y a la primera publicación en esta revista.

Analecta Veterinaria por Facultad de Ciencias Veterinarias se distribuye bajo una Licencia Creative Commons Atribución-NoComercial-SinDerivar 4.0 Internacional.